ACHIEVEMENTS
![]()
ACHIEVEMENTS研究成果
-

- ACHIEVEMENTS
- 自然が見出していない未踏のタンパク質トポロジー ー 新規αβ型トポロジーを持つタンパク質分子のデザイン ー
プレスリリース
2023.07.12
自然が見出していない未踏のタンパク質トポロジー ー 新規αβ型トポロジーを持つタンパク質分子のデザイン ー
自然科学研究機構 生命創成探究センター (ExCELLS)/分子科学研究所 協奏分子システム研究センターの、南慎太朗特任研究員(当時)、古賀(巽)理恵特任研究員(現:大阪大学蛋白質研究所助教)、古賀信康教授(兼任)(現:大阪大学蛋白質研究所教授)は、理化学研究所 生命機能科学研究センターの小林直宏上級研究員、長島敏雄上級テクニカルスタッフ、名古屋大学大学院工学研究科の千見寺浄慈助教、大阪大学蛋白質研究所の杉木俊彦特任助教(現:北里大学薬学部准教授)、藤原敏道大阪大学名誉教授と共同で、自然が見出していない未踏の折り畳み可能なタンパク質αβ型トポロジー*1が、現在自然界で発見されているトポロジー約400個よりも遥かに多く、少なくとも約1万個存在していることを世界で初めて理論と実験両方を用いて明らかにしました。この研究結果は、地球上の生命は、偶然発見したタンパク質トポロジーを繰り返し利用していることを示唆しています。また、タンパク質分子はその立体構造に基づいて機能を発現することから、新規トポロジーを持つタンパク質分子をデザインすることで、医療や産業に貢献する新規機能性タンパク質の創出に繋がると期待されます。
本研究成果は、国際科学雑誌 「Nature Structural and Molecular Biology」 (2023年7月3日付) に掲載されました。
論文情報
雑誌名: Nature Structural and Molecular Biology
論文名: Exploration of novel αβ-protein folds through de novo design
著者: Shintaro Minami†, Naohiro Kobayashi†, Toshihiko Sugiki, Toshio Nagashima, Toshimichi Fujiwara, Rie Tatsumi-Koga, George Chikenji, Nobuyasu Koga* (†共同筆頭著者、*責任著者)
DOI: 10.1038/s41594-023-01029-0
掲載URL: https://www.nature.com/articles/s41594-023-01029-0
1.発表のポイント
1. (本研究成果のハイライト)自然が見出していない折り畳み可能なタンパク質αβ型トポロジーの数を、世界で初めて理論と実験両方を用いて見積もった。
2. (本研究で得られた知見)「タンパク質分子が折り畳み可能なトポロジーを予測するルール」と「タンパク質分子をゼロからデザインする技術」を用いることで、現在自然界で発見されているαβ型トポロジー(400個)よりも、遥かに多くの新規トポロジー(少なくとも約1万個)が存在することを明らかにした。
3. (本研究の成果により期待される波及効果)新規トポロジーを持つタンパク質分子をデザインすることで、医療や産業に貢献する新規機能性タンパク質の創出に繋がることが期待される。
2.背景
生命現象を司るタンパク質分子は、アミノ酸配列に従って特定の立体構造を形成し、その立体構造をもとに機能を発揮します。タンパク質分子のアミノ酸配列空間*2は広大であり、たとえば100残基のタンパク質でも約10130通りの組み合わせが存在します。地球上に存在する生物が1000万種類あり、それぞれが10万個の固有の遺伝子を持っていると仮定すると、アミノ酸配列の総数は約1012通りとなりますが、これは10130に比べて非常に小さい数です。つまり、自然によって探索されていないタンパク質が膨大な数存在していると想像されます。
一方で、毎年1万個以上のタンパク質構造が実験的に解明されていますが、タンパク質のαヘリックスやβストランドの2次構造の配置とその連結方法で表されるトポロジー(フォールドとも呼ばれる)(図1)の観点から構造を見てみると、最近では新しいトポロジーが発見されることは非常に稀であることが知られています。タンパク質が折り畳み可能なトポロジーは、自然によってほぼ探索され尽くされたのか、それとも自然が未だに探索していない折り畳み可能なフォールドがまだ多く存在するのかについて、従来の研究では理論的な考察がされてきましたが、実験的な検証はされてきませんでした。
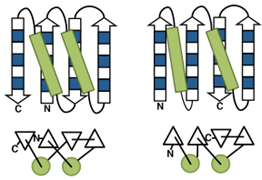
図1. (トポロジーとは?) αヘリックス(上:緑の長方形、下:緑の丸)やβストランド(上:青と白の四角形、下:白の三角形)の2次構造の空間上の配置と、ループの連結の仕方。図の左のトポロジーと右のトポロジーは異なる。トポロジーは、フォールドと呼ばれることもある。タンパク質科学以外でもトポロジーという言葉が用いられるが、ここでの定義とは異なることに注意。
3.本研究の内容
研究チームはまず、物理化学とタンパク質構造データに基づいて、タンパク質分子が折り畳み可能なトポロジーを理論的に見分けるルールを考案しました(図2)。次に、これらのルールを用いることで、現在のタンパク質構造データバンク*3に存在しないが折り畳み可能と考えられる4-8本のβストランドからなるβシートを持つ新規αβ型トポロジー(合計12,356個)を予測しました(図3)。さらに、これらのうち4本ストランドからなるβシートを持ち、結び目を形成するトポロジー*4を含む新規トポロジー8種類全てについて、これらのトポロジーのタンパク質を、研究チームが開発している「タンパク質分子を主鎖を含めてゼロからデザインする技術」を用い計算機でデザインしました。計算機でデザインしたタンパク質の折り畳み能を実験で調べたところ、これら全てのトポロジーのデザインについて計算機でデザインした通りの構造に折り畳んでいることが判明しました(図4)。この結果は、約1万個もの未踏の折り畳み可能なαβ型トポロジーが存在することを示しており、自然界に観測されるαβ型トポロジーの数は約400個であることを踏まえると、多くの折り畳み可能なトポロジーが自然に探索されず存在していると考えられます。
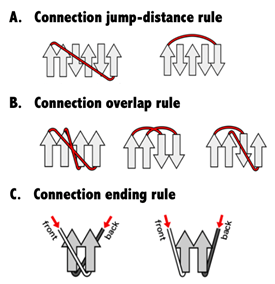
図2. (折り畳み可能なαβ型トポロジーを見分けるルール) A. βストランド間を連結する領域(赤)が他のβストランドを何本も跨がないこと。 B. βストランド間を連結する領域(赤)が交差しないこと。 C. 隣り合うβストランドに接続するループが異なるβシート面に存在しないこと。これらのルールを破っているものを上図で示した。本研究では、これらのルールを破っていないトポロジーを折り畳み可能なトポロジーとした。
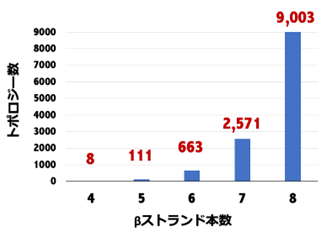
図3. (ルールで予測した折り畳み可能な新規αβ型トポロジーの数)
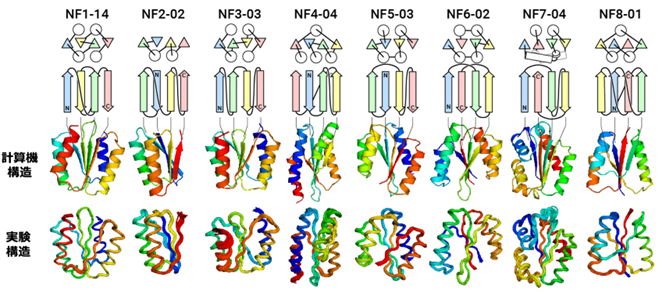
図4. (計算機でデザインした新規フォールドタンパク質) 上段からタンパク質名、トポロジー図(丸はαヘリックス、三角および矢印はβストランドを表す)、計算機でデザインした構造、実験で決定した構造を示す。計算機構造と実験構造は、よく一致する。NF8-01のトポロジーは結び目を形成する*4。
4.本研究の発見の意義
これらの研究結果から、タンパク質の構造と進化について以下のような仮説が考えられます。まず、生物進化の時間は現時点であまりにも短いため、全ての可能なトポロジーが探索されていない可能性です。他には、地球上の生命は共通の祖先から派生しており、この祖先関係により自然界で見られるタンパク質トポロジーに偏りがあるというものです。つまり、タンパク質は特定のタンパク質トポロジーを繰り返し再利用しながら、異なる機能を発現するように進化してきた可能性があります。もし、地球外に生命が存在しているとしたら、それらは他のタンパク質トポロジーセットを利用しているかもしれません。
5.今後の研究の展望
タンパク質は様々な機能を持つことで知られていますが、これらの多様な機能は、タンパク質の立体構造の多様性に基づいて生み出されています。今回の研究では、約1万個もの折り畳み可能なαβ型トポロジーが自然界で観測されず存在することが示されました。これら新規トポロジーを持つタンパク質をデザインすることで、さらなる構造の多様性を生み出せる可能性があり、医療や産業に貢献するような機能性タンパク質分子の開発が期待されます。
用語解説
※1 αβ型トポロジー:
βシートとαヘリックスからなるαβ型タンパク質において、αヘリックスやβストランドの2次構造要素の配置とその連結方法を表したもの。図1参照。
※2 タンパク質分子のアミノ酸配列空間:
タンパク質分子のアミノ酸は20種類からなるので、たとえば100残基のタンパク質の場合、アミノ酸配列空間は20100 10130通りの組み合わせからなります。
※3 タンパク質構造データバンク(PDB):
実験的に決定されたタンパク質構造が登録されているデータベース。
※4 結び目を形成するトポロジー:
タンパク質のN末端、C末端を持って引っ張ったときに結び目を形成するようなトポロジーのこと。
研究サポート
本研究は、日本医療研究開発機構(AMED)創薬等ライフサイエンス研究支援基盤事業(BINDS) 創薬等ライフサイエンス研究のための相関構造解析プラットフォームによる支援と高度化(創薬等ライフサイエンス研究のための多階層構造生命科学解析技術の支援と高度化) (JP19am0101072)、理研NMR研究基盤、自然科学研究機構アストロバイオロジーセンタープロジェクト (AB291007; 古賀信康)、科研費(15H05592; 古賀信康、18H05420; 古賀信康、18K06152; 小林直宏、19H03166; 千見寺浄慈、古賀信康)、JST未来社会創造事業(JPMJMI17A2; 小林直宏)、AMED BINDS(JP20am0101111; 千見寺浄慈)、科研費特別研究員(17J02339; 南慎太朗)、自然科学研究機構 岡崎共通研究施設 計算科学研究センター(22-IMS-C188, 21-IMS-C174, 20-IMS-C157, 19-IMS-C175, 18-IMS-C155, 17-IMS-C147)、分子科学研究所 機器センター、基礎生物学研究所 生物機能情報分析室、等の支援を受けて実施されました。
【蛋白質研究所】研究者紹介:古賀 信康教授、巽 理恵助教、藤原 敏道名誉教授

左より:古賀教授、巽助教
―ひと言コメントをお願いいたします―
新規トポロジーを持つタンパク質を人工設計するにあたって、特に結び目を形成するトポロジーのタンパク質を創ることは難しいだろうと思っていたため、NMRで構造が解かれ実際に創れたことが分かったときはとても驚きました。予断なく何でもやってみることが重要だと改めて思いました。
また、自然が見出していないトポロジーが少なくとも1万個以上もあることが分かったのも予想外なことでした。 現在我々が見ている自然の姿は、物理化学的に必然の部分もあると思うのですが、 意外と多くは偶然の結果で、他にも解はあったのかもしれません。
この研究は多くの方々の協力のもと実施することができました。ありがとうございました。


藤原名誉教授
―ひと言コメントをお願いいたします―
これまで蛋白研では、NMRによるタンパク質構造の効率的な解析法を整備してきました。
この方法が、その特徴を生かして本研究で役に立ちうれしく思います。






